想象一下,如果你的Google搜索引擎突然告诉你:“抱歉,互联网太大了,我只能帮你搜索其中1%的内容。”你会作何感想?是恐慌,还是无奈?
这听起来像天方夜谭,但这恰恰是过去十几年里,全世界微生物学家和流行病学家每天都在面对的窘境。他们赖以生存的基因“搜索引擎”——一个名叫BLAST的传奇工具——正被一场史无前例的数据海啸所淹没。
而今天,我们要讲的,就是一个关于如何在这场数据海啸中,重建一座能导航整个基因世界的“灯塔”的故事。
第一幕:昔日王者与数据海啸
在生物信息学的世界里,BLAST(Basic Local Alignment Search Tool)就是神。自上世纪90年代诞生以来,它就像基因世界的Google,科学家们想知道一段新发现的DNA序列“是谁”、“从哪来”、“有什么亲戚”,都会把它扔进BLAST里搜一下。几十年来,它支撑起了无数诺贝尔奖级别的发现。
但问题是,我们制造数据的能力,已经远远超过了我们分析数据的能力。
随着基因测序技术的成本雪崩式下降,全球的实验室每天都在向公共数据库上传海量的细菌基因组数据。这个数据库的规模,正以一种近乎失控的指数级速度膨胀。截止今天,我们已经拥有了数百万个细菌的完整基因“蓝图”。
这是一个巨大的宝藏,但也成了一场灾难。
面对这个比过去庞大成千上万倍的“基因互联网”,老国王BLAST力不从心了。它就像一个试图用手抄方式整理全世界所有图书的图书管理员,早已不堪重负。论文中一个令人震惊的事实是:如今,BLAST的网络版本能够搜索的细菌基因组,只占公开数据的极小一部分,而且这个比例还在“指数级下降”。
我们拥有了前所未有的生命数据,却几乎失去了全局搜索它的能力。这就好比,一个医生在病人身上发现了一种新的抗生素耐药基因,却无法快速知道它是否已在全球其他地方悄然流传;或者,疾控专家面对一场神秘的食物中毒,却无法在海量数据库中迅速锁定病毒的“亲戚”,找到源头。
整个科学界,都迫切需要一个全新的搜索引擎。
第二幕:困境中的“灯塔”建造者
故事的主角,是一群身处数据风暴中心的人。这篇论文的核心作者来自欧洲分子生物学实验室(EMBL-EBI)等机构,这里是全世界最大的生物数据存储和维护中心之一。他们不是在解决一个别人的问题,他们就是在解决“自己家仓库着火”的问题。
每天看着潮水般涌入的基因数据,和日益“卡顿”的分析工具,他们比任何人都更清楚,必须做点什么。
传统的搜索方法,无外乎两种思路。一种是“暴力搜索”,就像警察挨家挨户地毯式排查,把你的基因序列和数据库里数百万个基因组的每一段都比对一遍。这在数据量小的时候还行,现在这么干,可能你博士都毕业了,结果还没跑出来。
另一种是“关键词索引”,比如BLAST。它会把基因序列打散成一个个短小的“词汇”(比如28个字母长的DNA片段),然后为整个数据库建立一个庞大的“关键词索引”。搜索时,先匹配关键词,再延伸比对。但当数据库里有几百万本书(基因组),每本书都充满了各种独特的词汇时,这个“索引”本身就变得比图书馆还大,构建和查询都成了噩梦。
是时候跳出这个框架了。
第三幕:“Aha!”时刻与天才设计
这篇论文的灵魂,一个名为LexicMap的新工具,带来了一个极其聪明的解决方案。它的核心思想,可以用一个“万能钥匙”的比喻来理解。
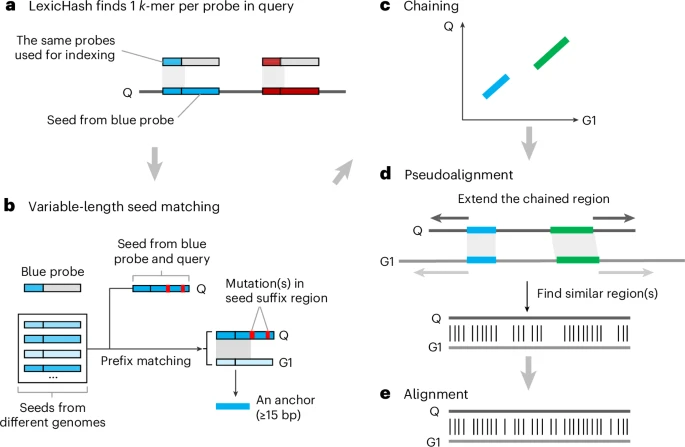
如果你想知道城里每栋房子里所有锁的型号,你不需要把每一把锁都拆下来研究。你只需要设计一套“万能钥匙胚”,比如20000个。这套钥匙胚被设计得非常巧妙,足以覆盖世界上所有主流的锁芯结构。
然后,你拿着这套钥匙胚,去每一栋房子里,为每一把锁都找到匹配度最高的那个钥匙胚,然后在那个胚上刻下这把锁独特的齿痕,并记录下它的位置。这些刻好的钥匙,就是这栋房子的“种子钥匙”。
LexicMap就是这么干的:
-
创造“万能钥匙胚”(Probes): 科学家们没有去索引数据库里数以万亿计的DNA片段,而是先巧妙地生成了一个固定的、可控的“模板库”,包含了20000个精心挑选的31个字母长的DNA短序列。这20000个模板,就像那套“万能钥匙胚”,它们的前7个字母,就足以覆盖所有可能的DNA“开头”。
-
为每个基因组生成“种子钥匙”(Seeds): 接下来,LexicMap拿着这20000个“模板”,去扫描数据库里的每一个细菌基因组。对于每个模板,它都会在基因组里找到一个与自己“长得最像”(前缀匹配最长)的DNA片段,并把它记录下来,称之为“种子”(Seed)。这样一来,每个几百万碱基长的基因组,就被浓缩成了20000个有代表性的“种子”坐标。
-
最关键的保证:“无沙漠”设计: 这是最天才的一步。为了防止某个关键基因恰好藏在一个没有任何“种子”的区域(论文里称之为“种子沙漠”),LexicMap有一个强制保证:在基因组的任何一个250个字母长度的窗口内,都必须有好几个“种子”存在。 如果发现有“沙漠”,它会立刻在该区域“人工播种”,确保万无一失。这就像确保城市地图上,每条街道都必须有几个摄像头一样,让任何目标都无处遁形。
现在,搜索变得异常简单。当你有一段新的基因序列(比如那个耐药基因)想要查询时,LexicMap先用同样的方法为你的序列生成20000个“种子”。然后,它要做的,就是一个纯粹的匹配游戏:看看数据库里哪些基因组的“种子”,和你的“种子”是对得上的。
一旦匹配上,就意味着你的基因序列很可能存在于那个细菌中。整个过程,从地毯式搜索,变成了一场高效的、基于“代表点”的匹配游戏。
更妙的是,这种匹配允许一定的模糊性。由于它只关心“前缀”或“后缀”是否相似,即使基因发生了一点点突变,就像钥匙稍微有点磨损,它依然能够识别出来,这让它在真实世界的应用中异常强大。
第四幕:王者归来,所以呢?
那么,这个全新的“基因Google”表现如何?
答案是:令人瞠目结舌。
论文中的测试结果(图4)就像一部好莱坞大片的爽文结局。当数据库的规模扩大到一百万个基因组时:
- 速度: LexicMap比第二快的工具快了3倍,比其他主流工具快了几十甚至上百倍。过去需要数小时甚至数天才能完成的搜索,现在,在你泡杯咖啡的时间里就完成了。
- 内存: 它对电脑内存的占用,低到不可思议。在百万基因组级别,它只用了6.2GB内存,而它的竞争对手们,一个需要717GB,另一个需要85GB。这意味着,过去只有超算中心才能跑的分析,现在一个普通的实验室服务器,甚至一台高性能笔记本电脑就能胜任。
- 准确性: 在如此巨大的性能优势下,它的准确率与现有最顶尖的工具相比,不相上下。
这不仅仅是一个工具的迭代,这是一次“范式转移”。它为所有生命科学家重新开启了一扇门。
所以,这对我们普通人意味着什么?
-
狙击超级细菌: 医院里出现了一种耐药性极强的“超级细菌”。医生可以立刻将它的耐药基因序列上传,用LexicMap在几分钟内扫描全球所有已知细菌基因组。我们可以立刻知道:这个耐药基因是全新的吗?还是已经在某个国家的某个农场里出现过?它的传播路径是怎样的?这种近乎实时的全球监控能力,是过去无法想象的。
-
秒速追溯疫情源头: 还记得追查新冠病毒源头的艰难吗?未来,当新的传染病出现时,科学家可以把病毒基因序列扔进LexicMap,在全球数百万的病毒和细菌数据库中搜索,快速找到它的近亲,分析其演化路径,这可能将疫情溯源的时间从数月缩短到数小时。
-
解锁生命的黑箱: 一个生态学家在深海火山口发现了一种奇特的微生物,它体内有个前所未见的基因。这个基因是干什么的?利用LexicMap,他可以搜索整个生命之树,看看有没有其他生物拥有类似的基因片段。如果发现这个基因的远房亲戚们都参与了某种抗压或能量代谢,这就为解开新基因的功能提供了决定性的线索。
就像BLAST在30年前彻底改变了生物学一样,LexicMap的出现,标志着我们终于拥有了与这个“大数据时代”相匹配的导航能力。
我们曾一度迷失在自己创造的知识海洋里,但现在, 一群不甘被数据淹没的科学家的奇思妙想,我们再次扬帆起航。一个全新的、充满无限可能的生物大发现时代,或许正悄然拉开序幕。